Pythonを使用したデータ解析入門01
どうも、ブログのサブタイトル通り更新をサボっていた有末です。
僕は生物学を専攻している関係上Pythonは主に研究データの解析に使用しています。本記事では懐石に関して今まで試行錯誤して得てきたノウハウを共有したいと思います。主に生データのグラフ化について書いていきます。タイトルに01とかつけてますが徒然なるままに更新されないブログなので次回作にはあまり期待しないでください。
前提
本記事では以下の条件を前提としています。例の通りWindowsは対象外ですのでGoogle先生に聞くなどして各種必要ライブラリのインストールなどを行なってください。なお下記で使用しているpipはWindowsに対応していなかったと思うので代わりにeasy_installを使用することになると思います。
なお下記例において%から始まる行はターミナルでの作業を表しています。ターミナル作業に関してわからないかたは下記Webページを参照してください。
pip のインストール
Pythonにはpipと呼ばれるパッケージ管理ソフトがあるのでこれを利用します。僕の場合はPythonを他の開発などにも使用しているためpythonbrewというソフトでバージョニングを行なっていますが、研究結果の解析程度であればOSにバンドルされているPythonで問題ないでしょう。下記ではOSバンドルバージョンを利用する例を紹介します。
Debian系OS(Ubuntu, Debian)の場合
% sudo apt-get install python-pip
Fedora, CentOS, RedHatの場合
% sudo yum install python-pip
Mac OS Xの場合
Homebrewがインストールされていることが前提です。
% echo "export PATH=/usr/local/bin:$PATH" >> ~/.bashrc
% echo "export PATH=/usr/local/share/python:$PATH" >> ~/.bashrc
% brew install python --framework
% /usr/local/share/python/easy_install pip
詳しい情報はMac OS XにPythonをインストールするに書かれていたので参照してください。
他必要ライブラリのインストール
pipさえインストールしてしまえば残りのインストールは簡単です……と言えれば良いのですが実はnumpy, scipyなどは純Pythonでは無いためpipを使用したインストールは上級向けになります(コンパイルという作業が発生するため初心者では太刀打ちできない)。したがってmpltools以外はOSバンドルバージョンをインストールするのが最も簡単な方法になります。
Debian系OS(Ubuntu, Debian)の場合
% sudo apt-get install python-numpy python-scipy python-matplotlib
% sudo pip install mpltools
Fedora, CentOS, RedHatの場合
% sudo yum install python-numpy python-scipy python-matplotlib
% sudo pip install mpltools
Mac OS Xの場合
numpyに関してはpipで行けるようですがscipyはFortranが必要など条件が様々あるようです。下記に従ってください。また英語が得意な方は参照元を参考にしてください。
% pip install numpy
% brew install gfortran
% pip install scipy
% brew install pkg-config
% pip install matplotlib
% pip install mpltools
参照元: Install Python, NumPy, SciPy, and matplotlib on Mac OS X
XYデータのグラフ化
生データとして最も多いタイプがこのデータ形式になると思います。例えば下記はECFPというシアン色蛍光タンパク質の蛍光を波長をふって取得したデータになります。生データはこちら
445.0000 72.5479 446.0000 86.7363 447.0000 106.924 ... 649.0000 9.14575 650.0000 11.7931
このようにXデータ(波長)とYデータ(蛍光強度)があるタイプをここでは便宜上XYデータと呼ぶことにします。XYデータでは単純に単純に各点をプロットすればよいため下記プログラムでグラフが描画できます(生データファイルをraw.datとして同フォルダ内に保存していると仮定しています)。
上記プログラムで作成されたグラフ(を表示しているウィンドウ)が以下になります。
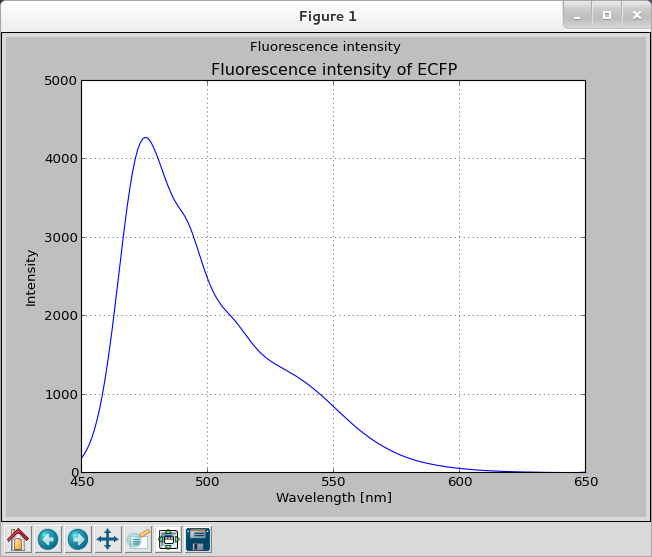
なお上記プログラムの注意事項として、ソースコード中で日本語を使用しているためコード上部にある# vim: set fileencoding=utf-8 :の表記がない場合はエラーになります。このようにソースコード中で日本語を使用する場合はソースコードの文字コードを指定してください。僕はVimmerなのでVim形式で指定していますが、通常# -*- coding: utf-8 -*-のようにEmacs形式で指定することが多いようです。魔法の言葉だと思い常に指定するようにしておいてください。
XYデータの相対値をグラフ化
別データと比較する場合何%減少したか?などの相対値が重要になると思います。下記グラフはある条件下における蛍光強度変化を測定したものです。別タンパク質や別環境と比較するために初期条件からどの程度変化したのかを表す相対値でプロットしています。
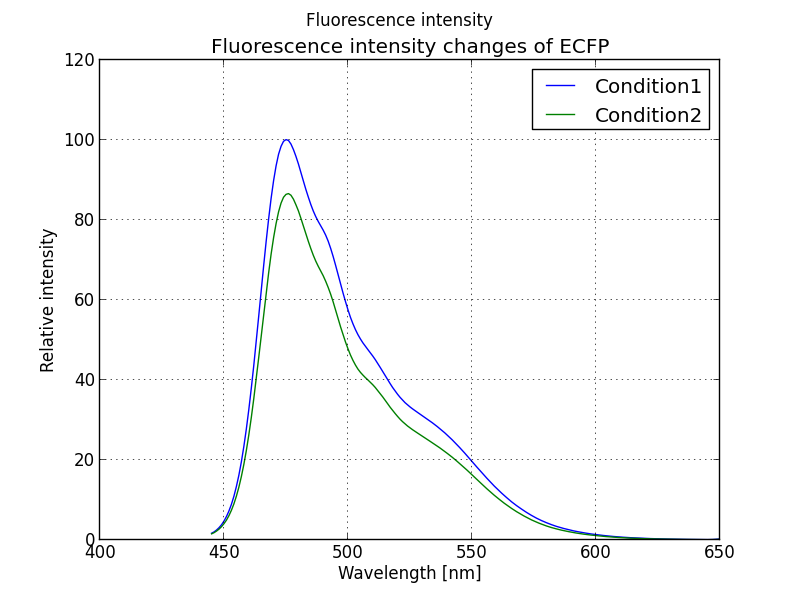
このような相対値をグラフ化するには先のプログラムに少し修正を加えます。下記を参照してください。
XYデータの条件におけるピーク変化量をグラフ化する
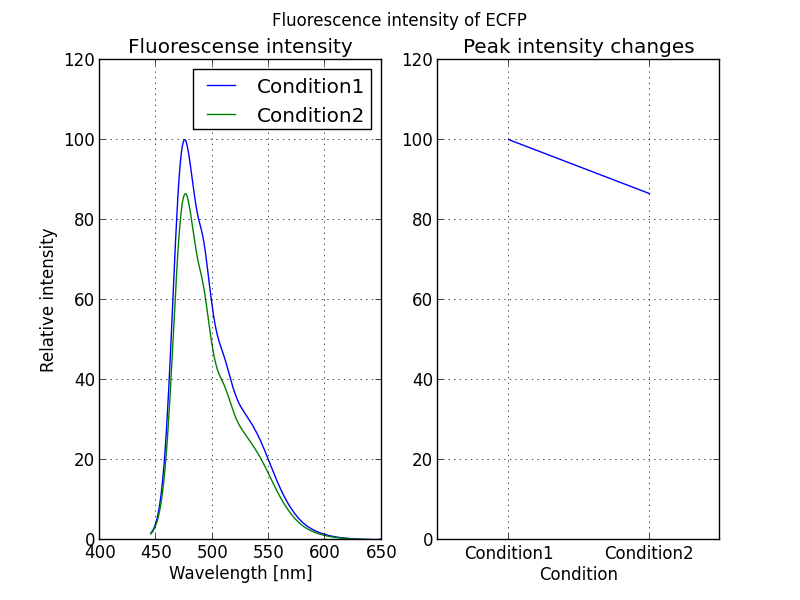
先のグラフではXYデータが条件変化によりどのように変化するのかの概要をつかむことはできましたがピークの値がどの程度変化したのか?を知るには不適切なグラフとなります。なので下記グラフのように横軸に条件、縦軸にピークの値をとったグラフを作りましょう。
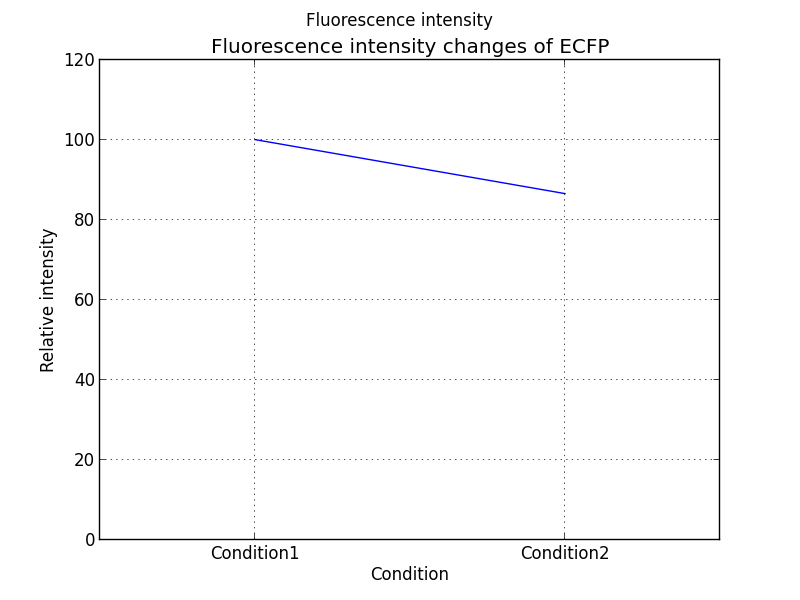
先のプログラムに更に改良を加えるとこのようなグラフを作ることができます。下記を参照してください。
相対値グラフとピーク変化グラフを一つにまとめる。
最後に作成した二つのグラフを一つにまとめましょう。もちろんWordなどを利用してまとめても良いのですがmatplotlibにはsubplotと呼ばれる機能があり、複数のグラフをまとめることができるので今回はこれを利用します。下記プログラムを実行するとこの記事の扉絵のようなグラフが出来上がります。
最後に
個数データなどは対応できないのでヒストグラムを用いてください。Pythonでヒストグラムを書いてガウシアンフィッティングでクラスタリングするサンプルを昔書いたのでそちらも参考にしてください。どんぐりの背比べ(混合ガウスモデルによるクラスタリング)